Historique de l'étude des bactériophages
Un siècle de recherche sur les bactériophages
Comme souvent dans l’histoire des sciences, la découverte des virus « mangeurs de bactéries » (i.e. « bactériophages ») s’est déroulée en plusieurs phases dans le prolongement des premiers balbutiements de la virologie. En effet, dès 1881, Louis Pasteur (1822-1895) montre que l’agent infectieux de la rage, invisible au microscope, passe librement à travers un filtre Chamberland qui est un filtre en porcelaine poreuse retenant les particules de taille visible au microscope optique (pores de diamètre < 300 nm), et est transmissible. En 1892, Dmitri Ivanovski (1864-1920) découvre le virus de la mosaïque du tabac : la sève d’un plant malade filtrée à travers un filtre Chamberland contient un agent invisible capable de causer la maladie lorsqu’il est mis en contact avec un plant sain. On estime aujourd’hui que le bactériologiste britannique H. Hankin (1865-1939) est le premier auteur à avoir mis en évidence l’existence d’un agent bactéricide présent dans l’eau (Figure 1). Travaillant alors sur le bacille cholérique, au sein d’un laboratoire gouvernemental en Inde, il décrit dans une publication princeps en 1896 la destruction rapide du bacille lorsqu’une culture de celui-ci est mise en contact avec de l’eau du Gange et de la Jumna [1,2]. Il démontre que cet agent est capable de traverser un filtre Chamberland et est détruit par l’autoclavage. Par contre l’eau récupérée des puits ne possède pas cette capacité destructrice de bacilles. Même si à cette époque Hankin est incapable d’appréhender le phénomène avec plus de précision, l’existence d’un agent antibactérien très efficace, présent naturellement dans des eaux qui contiennent elles-mêmes la bactérie sensible est bien avérée.
Ces découvertes concomitantes sont importantes sur un plan général : elles révèlent expérimentalement l’existence de nouveaux agents infectieux qui s’attaquent à des cellules humaines dans le cas de la rage, à des plantes dans le cas du virus de la mosaïque du tabac ou à des bactéries et dont la taille est inférieure à celle des bactéries puisqu’ils passent à travers des filtres retenant ces dernières. Frederick Twort (1877-1950) fut le second bactériologiste à aborder directement les agents antibactériens invisibles. Il confirme leur caractère ultra-filtrable et leur inactivation par la chaleur et montre également que ces particules ne se développent plus dès lors que l’échantillon est exempt de bactéries comme décrit dans sa publication de 1915 [3]. Deux ans plus tard, en 1917, le chercheur franco-canadien Félix d’Hérelle (1873-1949), alors qu’il travaille à l’Institut Pasteur, pose certains fondamentaux de virologie: il décrit l’isolement et la spécificité de ces agents « antagonistes » ainsi que la formation de plages de lyse sur un tapis bactérien [4]. Il proposera l’appellation « bactériophage », du grec ancien bakterion (petit bâton) et phágos (mangeur), et sera le premier à les utiliser dans une optique thérapeutique, notamment dans le contexte de la lutte contre les dysenteries bacillaires provoquées par Shigella, donnant ainsi naissance à la phagothérapie [5,6]. A sa suite, son collaborateur Georges Eliava de retour à Tbilissi, capitale de la Géorgie, fonde le centre thérapeutique et de recherche qui porte encore aujourd’hui son nom. L’article de revue suivant traitera plus précisément des applications des bactériophages, dont les applications thérapeutiques.
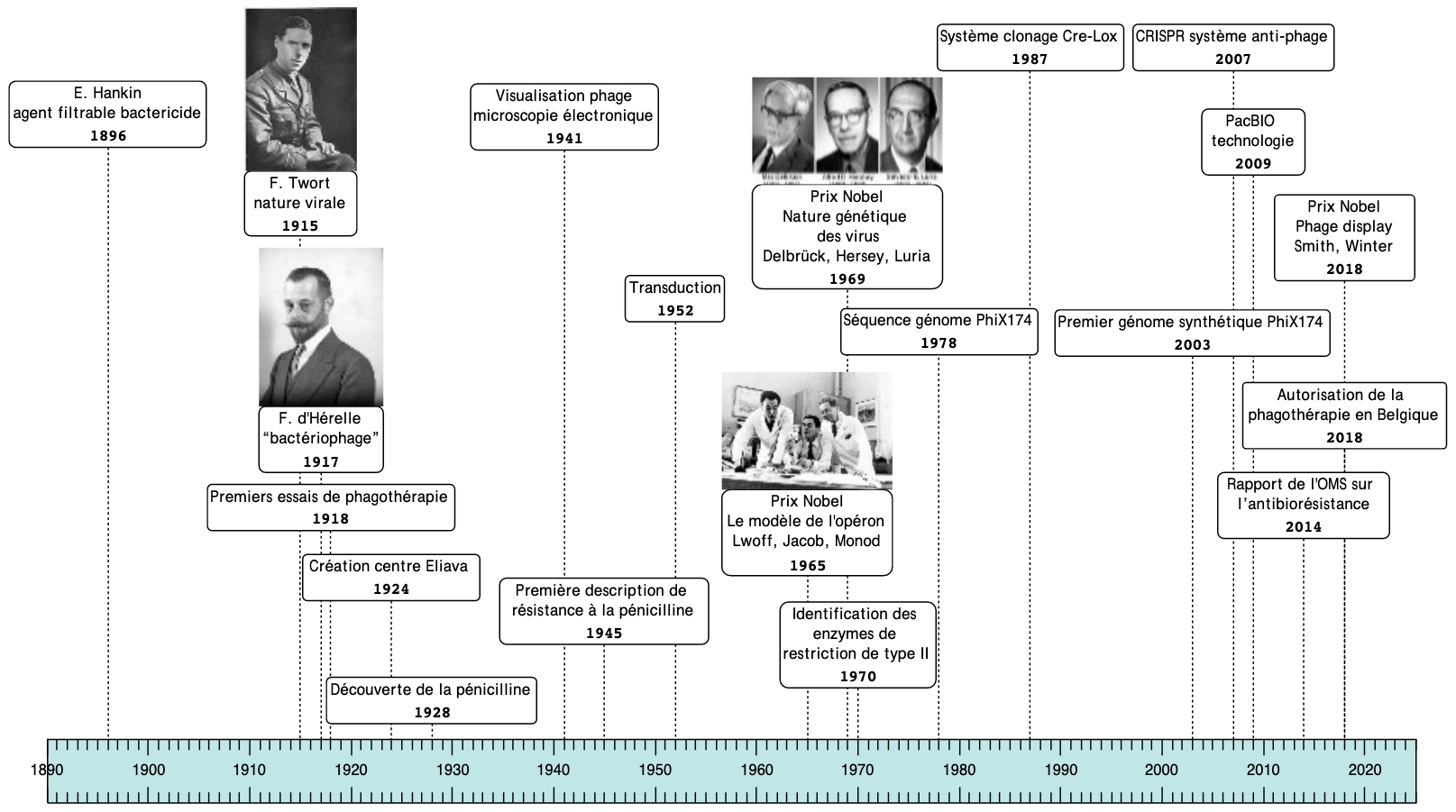
Figure 1. Les grandes étapes des cent premières années de recherche sur les bactériophages.
Le rôle des bactériophages dans la naissance de la biologie moléculaire C’est dans la deuxième moitié des années 1930 que le physicien allemand Max Delbrück (1906-1981) rejoignit le déjà très renommé California Institute of Technology pour y débuter des travaux permettant de faire un lien entre physique et biologie, à une époque où des termes tels que gène ou protéine étaient encore mal appréhendés. C’est au contact d’Emory L. Ellis (1906-2003) qu’il s’initie à la biologie des bactériophages [7]. Puis, en collaboration avec Salvador Luria (1912-1991) et Alfred D. Hershey (1908-1997) il va poser les bases du « phage group » qui sera à l’origine de nombreuses études ayant pour objet les bactériophages, sur lesquelles reposent les fondations de la biologie moléculaire [8]. En étudiant le cycle lytique du bactériophage T2, ils ont établi les bases de la génétique et de l’évolution moléculaire avec la démonstration de mutations apparaissant au hasard au cours du temps. Ces travaux ont permis de rejeter l’hypothèse selon laquelle les bactéries résistantes n’émergeaient qu’en présence du virus [9]. En 1952, les travaux menés par Alfred Hershey et Martha Chase ont démontré que le support physique de l’information de l’hérédité est bien l’ADN et non les protéines comme on le croyait. En effet, le marquage individuel de l’ADN et des protéines lors d’une infection d’E. coli par le phage T2 à l’aide d’isotopes radioactifs a permis d’observer que seul l’ADN était transféré dans le cytoplasme de la cellule infectée, tandis que les protéines formaient une structure protégeant l’ADN et responsable de la fixation du virus à la bactérie [10]. Ces travaux primordiaux ont été reconnus et récompensés en 1969, par l’attribution du prix Nobel de Physiologie ou Médecine, aux biologistes Max Delbrück, Alfred Hersey et Salvador Luria pour leur découverte du « mécanisme de réplication et la structure génétique des virus ». Cette distinction s’inscrit dans un mouvement de reconnaissance de l’apport des travaux sur les bactériophages et fait suite à l’attribution du prix Nobel décerné aux pasteuriens André Lwoff (1902-1994), François Jacob (1920-2013) et Jacques Monod (1910-1976) en 1965, pour leurs études sur la lysogénie et la publication de l’article décrivant « le modèle de l’opéron » et le répresseur du bactériophage λ [11,12]. Le trio énonce ainsi les concepts de la structure d’un gène et de la régulation de son expression. Le phage λ, modèle du phage tempéré, découvert en 1950 par Ester Lederberg (1922-2006) a permis quant à lui de poser les bases de la lysogénie [13].
Dès 1941, Ernst Ruska (1906-1988), l’un des inventeurs du microscope électronique, capture des images de bactériophages, travail complété ensuite par S. Luria [14]. Les premiers essais de séquençage par Frederik Sanger (1918-2013) ciblèrent le bactériophage φX174 et conduisirent au décryptage du premier génome à ADN double brin [15] un an après celui du génome à ARN du phage MS2 [16]. Les contributions de l'étude des phages à la biologie moléculaire moderne sont très importantes et utilisées quotidiennement sans que l’on se rappelle parfois l’origine de ces techniques, qu’il s’agisse des systèmes de transcription in vitro utilisant la polymérase du bactériophage T7 ou des systèmes de clonage par recombinaison site-spécifique de type Cre-lox, λ ou ϕC31, qui permettent le clonage à haut débit et sans cicatrice [17–20]. A l’heure actuelle, l’une des dernières technologies de séquençage à haut débit (PacBio) utilise les propriétés de l’ADN polymérase du bactériophage φ29 dont la processivité très importante permet le séquençage de longs fragments d’ADN [21].
L’histoire de la biologie moléculaire est intimement liée aux découvertes réalisées sur la biologie des bactériophages ou de leurs interactions avec les bactéries. En particulier, les applications spectaculaires des systèmes anti-bactériophages, mieux connus sous les noms de ciseaux moléculaires ou CRISPR-Cas9, mais aussi des nouvelles découvertes sur l’immunité bactérienne, indiquent que les bactériophages vont continuer à jouer un rôle majeur dans les développements futurs de la biologie moléculaire [22].
Les cycles d’infection des bactériophages en résumé
L’immense majorité des bactériophages isolés à ce jour (> 95%), sont des bactériophages caudés, formés d’une capside protéique icosaédrique contenant une molécule d’ADN linéaire double brin connectée à une queue permettant le transfert du génome viral dans le cytoplasme se l’hôte [23,24]. D’autres groupes morphologiques se distinguent par l’absence de queues et comprennent des phages icosaédriques avec ou sans membrane lipidique, des phages filamenteux ou encore pléomorphes, contenant des génomes à ADN simple brin, à ARN simple ou double brin (Figure 2) [23–25].
Figure 2. Les principales familles de bactériophages
Comme tous les virus, les bactériophages sont des agents infectieux, parasites obligatoires dépourvus d’une machinerie de traduction qui ont besoin d’un hôte cellulaire pour se développer et produire une descendance (Figure 3). Dans cette revue, uniquement les aspects des relations phages-bactéries seront abordés, mais il faut noter que le site ViralZone (https://viralzone.expasy.org/) présente une vue d’ensemble de la conservation des processus biologiques et moléculaires communs à tous les virus, qu’ils infectent des cellules eucaryotes ou procaryotes.
La première étape de l’infection est la reconnaissance de la cellule hôte, qui est suivie de l’injection du matériel génétique dans le cytoplasme bactérien. Lors d’un cycle lytique productif, il y a réplication immédiate du génome, transcription et traduction des gènes viraux. L’orchestration des étapes est finement régulée et conduit à l’assemblage de nouvelles particules virales qui seront libérées par la lyse de la cellule sous l’action d’enzymes virales, les endolysines. Les bactériophages dits virulents sont uniquement capables de réaliser un cycle lytique, alors qu’une alternative s’offre aux bactériophages dits tempérés : la lysogénie. Dans ce cas, le génome phagique n’est pas répliqué de façon massive mais maintenu dans le cytoplasme de l´hôte, la plupart du temps sous forme intégrée dans le chromosome. L’intégration quand elle a lieu s’opère par recombinaison site-spécifique alors que d’autres prophages se maintiennent sous forme épisomale [26,27]. Contrairement au destin de mort cellulaire qui survient lorsque la bactérie est infectée par un bactériophage virulent, la bactérie lysogène survit et se divise normalement tout en permettant la transmission verticale du génome phagique. Cependant, en cas de stress de l’hôte, le prophage parvient à détourner les voies de signalisation du stress et peut alors redémarrer un cycle lytique par la réplication du génome ce qui aboutit à la dissémination des virions dans l’environnement (Figure 3) [28–30].
Figure 3 : Représentation schématique et simplifiée des cycles lytique et lysogénique. Adapté de [36].
Grâce à la démocratisation du séquençage génomique, la prévalence des prophages dans les génomes bactériens est établie et ils y constituent une source majeure de diversité génétique. Les prophages apportent à leurs hôtes des avantages sélectifs importants comme des facteurs d’adaptation à la fluctuation de l’environnement, et/ou des facteurs de virulence [31–33]. Ainsi, beaucoup de bactéries responsables de pathologies humaines (choléra, botulisme, shigellose, diphtérie, coqueluche…) produisent des toxines dont les gènes sont situés dans des prophages. Les bactéries correspondantes peuvent devenir avirulentes, ou perdre tout ou partie de leur caractère infectieux, lorsque le prophage est perdu [34]. D’autres fonctions plus subtiles sont codées par les prophages telles que des éléments de régulation génétique croisées entre gènes bactériens et de prophages [35]. De plus, dans de nombreux cas, les prophages résidents confèrent une résistance aux bactériophages qui tentent d’infecter la bactérie lysogène. Cette résistance implique différents mécanismes tels que la modification du récepteur bactérien ou la dégradation de l’ADN entrant [32]. L’ensemble des propriétés conférées par les prophages contribuent à la physiologie des populations bactériennes dans différents niches telle que l’intestin humain dans lequel la grande majorité des bactéries sont lysogènes et contiennent un ou plusieurs prophages [33]. Ces prophages sont parfois inactivés par des mutations les empêchant de s'exciser ou de s’induire apportant ainsi de façon permanente certaines fonctions qui sont cooptées par l'hôte bactérien.
La reconnaissance de l’hôte
L’étape de reconnaissance de l’hôte est cruciale et déterminante pour la suite de l’infection et varie selon le type d’hôte considéré (Gram+ ou Gram-, présence d’une capsule ou non, d’appendices extracellulaires ou non…). Chaque bactériophage reconnaît de manière très spécifique un ou plusieurs composants exposé(s) à la surface de l’hôte. La spécificité de cette interaction contraint le spectre d'hôte des bactériophages aux bactéries qui expriment le récepteur. L'interaction se fait via les protéines de liaison au récepteur du bactériophage (ou Receptor Binding Protein, RBP), situées à l’extrémité des fibres caudales et la nature des récepteurs varie selon les caractéristiques de l’enveloppe bactérienne [37]. Les bactéries qui possèdent une membrane externe (Gram négatif) exposent à la surface des lipopolysaccharide (LPS) qui sont communément utilisés comme récepteur initial [38] . Des protéines de membrane externe, telles que les porines, certains systèmes de sécrétion, les récepteurs de sidérophores et de vitamines, permettent un ancrage irréversible du bactériophage, aussi appelé adsorption, qui déclenche alors l’injection du génome dans la bactérie hôte [38]. D’autres structures extracellulaires telles que les capsules, les exopolysaccharides, les fibres ou les flagelles et pili peuvent également servir de récepteurs. De nombreux bactériophages disposent d’enzymes de dépolymérisation qui dégradent les motifs glucidiques pariétaux. Cette activité augmente la capacité de pénétration des bactériophages à travers les enveloppes complexes, facilitant ainsi l’accès à la membrane bactérienne où se situent les récepteurs protéiques [39]. Chez les bactéries à Gram positif, les acides téichoïques constituent les récepteurs primaires dont la nature et composition variées modifient l’adsorption des bactériophages à leur surface [40].
L’assemblage et la structure des particules phagiques
L’assemblage des bactériophages, comme celui de la plupart des virus, suit une séquence régulée et ordonnée d’interactions protéines/protéines et protéines/acides nucléiques. Les mécanismes moléculaires qui contrôlent cet assemblage ont été élucidés pour différents phages modèles grâce à des approches multiples combinant génétique, biochimie, analyses biophysiques sur particules isolées et biologie structurale. Les études liées à l’étude du cycle lytique du phage λ ont permis par ailleurs la découverte des chaperons moléculaires impliqués dans la réplication des génomes (DnaK/DnaJ) et l’assemblage des virions (GroES/GroEL) [41]. L’identification de gènes d’assemblage, la localisation dans les particules phagiques des protéines structurales par immuno-marquage et la résolution de leur structure atomique par RMN et cristallographie aux Rayons X et enfin, le recours aux techniques de pointe en matière de cryo-microscopie électronique ont permis d’obtenir des informations à très haute résolution sur l’architecture des bactériophages caudés [42]. Il est à noter que la cryo-microscopie électronique, développée dès les années 1980 par J. Dubochet et ses collaborateurs et particulièrement utilisée pour étudier l’organisation de l’ADN double brin dans les capsides de phages, lui a valu le Prix Nobel de Chimie en 2017 [43,44]. On distingue 3 familles de bactériophages caudés en fonction de la structure de leur queue : les myoviridae (queue contractile), les siphoviridae (queue flexible non contractile) et podoviridae (queue courte) (Figure 2). Chez les deux premiers groupes, la capside et la queue sont assemblées séparément et sont ensuite connectées pour former la particule infectieuse. Chez les podoviridae, les protéines de queue se fixent à la capside après son assemblage. Pour tous ces bactériophages, une procapside vide est d’abord formée qui contient un pore d’entrée formé par la protéine portale qui permet le passage de l’ADN au niveau d’un sommet unique de l’icosaèdre. Puis une terminase forme avec la portale un moteur moléculaire extrêmement puissant, qui transporte et condense l’ADN viral à l’intérieur de la capside en utilisant l’énergie d’hydrolyse de l’ATP [45,46]. Cette encapsidation s’accompagne d’une réorganisation coopérative de toutes les sous-unités de la protéine de capside induisant l’expansion de la capside qui acquiert alors sa taille définitive [47]. Cette étape se termine avec la fermeture de la portale par des protéines formant le connecteur ou site d’attachement de la queue [48] et la capside pleine peut être décorée ou renforcée par des protéines qui cimentent sa surface [49]. La capside mature est une structure extrêmement stable, capable de résister à une pression interne résultant de la très forte compaction de l’ADN double brin et pouvant atteindre 60 atm [50].
La construction de la queue, chez les siphoviridae et myoviridae, commence par l’assemblage du complexe basal qui contient les protéines de liaison aux récepteurs et constitue l’appareil de perforation de l’enveloppe bactérienne. Cette structure qui comprend seulement quelques dizaines de protéines chez la plupart des siphoviridae comme T5 ou TP901, peut être beaucoup plus complexe et contenir près de 150 copies d’une quinzaine de protéines différentes comme dans le cas du bactériophage T4 [51–53]. Il y a ensuite polymérisation hélicoïdale de la protéine majeure de queue autour d’une protéine centrale qui remplit le tube interne de la queue et agit comme un vernier pour en définir la longueur. Chez les myoviridae, la queue est entourée d’une protéine formant la gaine du système contractile [54]. L’assemblage de la queue s’achève à la fin de la polymérisation du tube caudal par la fixation de protéines de terminaison qui assurent la liaison avec le connecteur de la capside. Les bactériophages caudés constituent des exemples exceptionnels d’assemblages nucléoprotéiques de très grande masse moléculaire dépassant souvent la centaine de MDa. Les additions successives des différents composants protéiques à partir d’un complexe initiateur sont principalement contrôlées par des changements conformationnels et l’intervention des protéines chaperons de l’hôte qui stabilisent chaque état intermédiaire de l’assemblage. De manière remarquable, la stabilité des bactériophages est assurée par des interactions non covalentes, interactions hydrophobes, électrostatiques, liaisons hydrogène et présence de ponts salins [55]. Les progrès récents de la cryo-microscopie électronique et des méthodes de reconstruction tridimensionnelle des complexes macromoléculaires permettent maintenant de décrypter les processus d’assemblage et de déterminer la structure des bactériophages à l’échelle quasi atomique [56,57]. En utilisant ces techniques, combinées à la cristallographie aux rayons X, plusieurs équipes viennent de mettre à jour les processus dynamiques qui accompagnent la fixation du bactériophage sur son hôte. Ainsi les modifications structurales des protéines de queue induites par la liaison bactériophage/récepteur et qui déclenchent la libération du génome hors de la capside et son injection dans la bactérie viennent d’être révélées : mécanisme de déploiement des fibres du phage T4, réarrangement des protéines de la queue du bactériophage P22, rôle de protéine vernier du tube caudal du phage T5 dans la transmission du signal d’ouverture de la capside, et rôle des domaines des protéines du bactériophage Tuc2009 responsables de la fixation aux sucres de surface des lactobacilles [51–53,58]. Les techniques de cryo-tomographie électronique permettent également d’étudier les mécanismes de perforation de l’enveloppe bactérienne par les protéines caudales [57,59].
La structure et l’assemblage des virions de la famille des bactériophages filamenteux, moins nombreux que les bactériophages caudés, ont aussi été largement étudiés. L’exemple le plus connu est celui du bactériophage M13, qui forme un bâtonnet semi-flexible de 900 nm de long et 6 nm de diamètre. Il est constitué de 2700 copies d’une même protéine majeure qui forment une gaine de symétrie hélicoïdale autour d’un ADN circulaire simple brin et de quatre protéines minoritaires localisées à chaque extrémité. Ces bactériophages sont assemblés simultanément à leur sécrétion à travers l’enveloppe de leur hôte et ce processus a lieu sans lyse de la bactérie [42]. La possibilité de fusionner des séquences codant des peptides étrangers avec les gènes codant les protéines de structure des bactériophages filamenteux sans affecter leur repliement permet d’exposer ces peptides à la surface des bactériophages. La technique dite du « Phage Display » est actuellement très utilisée pour présenter des peptides aléatoires à la surface des bactériophages [60]. Leur tri basé sur leur capacité à se fixer à un ligand spécifique d’un des peptides exposés à leur surface suivi de leur amplification sélective, offre de nombreuses applications pour identifier et étudier les interactions protéine-protéine. Cette technologie a valu le Prix Nobel de Chimie à l’Américain George P. Smith et au Britannique Sir Gregory Winter en 2018 (Fig. 1).
L’ensemble de ces études structurales mettent en exergue l’importance d’étudier également les phages n’appartenant à l’ordre des caudovirales, pour lesquels moins d’espèces ont été identifiées. Les modes d’entrée et de lyse de ces phages n’est pas toujours élucidé et fait actuellement l’objet de nombreuses études. A la différence des phages caudés qui possèdent des protéines spécifiques capables de former un canal permettant le transfert de leur ADNdb à travers l’enveloppe bactérienne, les bactériophages dépourvus de queue utilisent des mécanismes d’entrée très variés pour internaliser leur génome ADN ou ARN dans la cellule hôte. L’exemple le mieux décrit chez les Tectiviridae est le phage PRD1, dont la capside entoure une membrane phospolipidique riche en protéines, qui protège un ADNdb linéaire. La pénétration de ce génome est canalisée par un tube lipidique qui se forme au niveau d’un sommet spécialisé de la capside icosaédrique [61]. Chez le phage PM2, de la famille des Corticoviridae, la membrane interne du phage est également impliquée dans l’internalisation de l’ADNdb circulaire, probablement en fusionnant avec la membrane externe de l’hôte [62]. Chez le microvirus X, les 10 copies de la protéine pilote H, dont la localisation dans la capside n’est pas connue avec précision, s’organisent pour former un tube qui canalise le transfert de l’ADNsb circulaire à travers l’enveloppe bactérienne [63]. Le mécanisme d’entrée des Plasmaviridae, dont le génome est constitué d’ADNdb circulaire surenroulé contenu dans une membrane protéolipidique, reste hypothétique. Ces phages pléomorphes infectent des bactéries dépourvues de paroi cellulaires comme les mycoplasmes. Leur mode de pénétration dans l’hôte pourrait impliquer la fusion de leur membrane avec la membrane cytoplasmique bactérienne [64]. Chez les Cystoviridae comme 6, dont une ARN polymérase ARN-dépendante, qui doit être internalisée avec l’ARN car elle est nécessaire à la réplication et transcription du génome viral dans la bactérie hôte Pseudomonas syringae. Lors de l’infection, 6 se fixe sur le pilus retractile de P. syringae, entrainant la fusion de la membrane virale avec la membrane externe bactérienne et permettant la libération de la nucléocapside dans le périplasme. Grâce à l’action d’une endopeptidase qui digère le peptidoglycane la nucléocapside accède à la membrane plasmique qui s’invagine pour l’enfermer dans une vésicule intracellulaire, comme lors de l’entrée par endocytose des virus eucaryotes [65]. D’autres phage se fixent aux pili bactériens pour initier l’entrée de leur génome, en particulier les phages filamenteux à ADNsb circulaire ou Inoviroidae tels que fd, f1 et M13 qui utilisent le pilus F de E. coli ainsi que le complexe macromoléculaire TolQRA de l’enveloppe bactérienne [64,66]. Les très petits phages à ARNsb de la famille des Leviviridae comme MS2 and Q, utilisent une protéine de maturation de la capside qui se fixe au pilus et induit le transfert de l’ARN dans le cytoplasme [64].
Les études structurales des bactériophages nous éclairent sur les propriétés fascinantes de ces « nanomachines » dont le programme d’assemblage, codé par un nombre de gènes restreint, contrôle avec une très grande fidélité la construction de particules formées de plusieurs centaines de protéines et dont la masse moléculaire peut dépasser 100 MDa [67]. L’intérêt de comprendre ces mécanismes est multiple, en particulier en biotechnologie. De nombreux projets de recherche se basent sur les données structurales pour produire des bactériophages modifiés ou des nanoparticules biocompatibles dérivées des bactériophages.
Impacts écologique et évolutif des bactériophages
Les bactériophages représentent les organismes les plus nombreux et les plus divers sur Terre [68]. En effet, on estime que les bactériophages (virulents et tempérés) sont présents partout où se trouvent leurs hôtes bactériens avec un ratio [bactériophages/bactéries] variable selon les environnements et les outils d’estimations. On trouve ainsi des bactériophages infectant les bactéries présentes dans les océans, les lacs d’eau douce ou salée, différents compartiments des vertébrés tels que l’intestin, la cavité buccale, la peau ou les poumons, ainsi que chez les insectes, notamment ceux hébergeant des bactéries symbiotiques [69,70]. On trouve aussi des bactériophages associés aux bactériomes (populations bactériennes associées à un écosystème particulier et composées de multiples espèces) des plantes que ce soit dans la rhizosphère, les racines ou les parties aériennes [71,72].
Les interactions entre les bactériophages et leurs hôtes relèvent clairement des interactions durables. Ainsi, les bactériophages virulents ont une interaction de type prédateur-proie, dans laquelle leur reproduction implique la mort cellulaire, alors que les bactériophages filamenteux ont des interactions parasitaires, puisqu'ils sont produits et secrétés sans mort cellulaire. Les bactériophages tempérés ont des interactions plus complexes. En phase lytique, ils se comportent comme des bactériophages virulents. En tant que prophages, ils ont des interactions parasitaires quand ils ont un coût pour la cellule, voire des interactions mutualistes quand les valeurs sélectives de la bactérie lysogénique et de son prophage sont mutuellement augmentées [73,74]. Les pressions de sélection exercées par les bactériophages sur les populations bactériennes contribuent à la diversification microbienne dans tous les environnements. Ceci participe par effets interposés à des changements plus larges qui influencent l’état sanitaire des organismes multicellulaires hébergeant des populations bactériennes [75]. De ce fait, les bactériophages ont également une influence sur les paramètres physico-chimiques des écosystèmes, le cycle du carbone et jusqu’au changement climatique de la planète [76]. Nous détaillons ci-dessous les pressions de sélection exercées par les bactériophages sur les populations bactériennes et les dynamiques écologiques et évolutives qui découlent de ces interactions.
Les bactériophages, ne pouvant pas se déplacer activement, dépendent entièrement de la probabilité de rencontre des bactéries par collision aléatoire. Ainsi leur capacité à infecter et à se reproduire est déterminée par leur capacité à rester infectieux dans l’environnement en raison de la stabilité et de la résistance de leur capside, mais aussi par la densité des bactéries sensibles : pour qu’un bactériophage puisse se multiplier, il est nécessaire que la densité de bactéries soit supérieure à une densité critique appelée « seuil de réplication ». En dessous de cette densité, la probabilité d’infection est trop faible et le bactériophage ne peut pas s’amplifier. En conséquence, les bactériophages associés aux espèces bactériennes les plus adaptées à un écosystème et qui dépassent donc un seuil critique, sont ceux qui se reproduisent le plus. Les phages sont donc responsables des cycles de renouvellement des populations bactériennes les plus abondantes et les mieux adaptées, un concept connu sous l’expression « tuer le vainqueur » [77,78].
Figure 4. Interaction « tuer le vainqueur » (source : [58]). Étape 1 : les bactéries sont au-dessous de la densité critique, si bien que malgré la densité élevée en bactériophages (environ 10 fois supérieure aux bactéries à la surface des océans), les bactéries ne sont pas infectées. L’écosystème est en état d’équilibre dynamique. Étape 2, une espèce bactérienne prolifère et dépasse le seuil de réplication permettant l’infection par les phages présents (vert). L’équilibre est modifié, l’espèce verte « gagne ». Étape 3, les bactériophages (verts) se multiplient sur cette espèce, qui lyse (vert clair, contour en pointillés) et s’effondre. Le gagnant est tué. Étape 4, les bactéries vertes retombent au-dessous du seuil de réplication. Le système revient à l’équilibre.
Malgré la popularité de ce concept, peu de démonstrations ont été rapportées dans le cadre de l’interaction entre les bactériophages et leurs hôtes. Ainsi, l’une de ces rares études a montré que les populations de Vibrio cholerae dans des environnements aquatiques oscillaient de manière anti-corrélée dans le temps avec certains bactériophages ciblant cette espèce [79]. De manière similaire, les cyanophages structurent la dynamique de population de leur communauté d'hôtes (les cyanobactéries Synechococcus et Prochlorococcus) tout en ayant un impact sur le cycle biogéochimique. Ainsi, le nombre de cyanophages augmente pendant les mois d’été propices au développement de leurs hôtes [80]. A plus grande distance dans la chaine alimentaire, la prédation par les bactériophages peut conduire à une limitation de la ressource en bactéries, source alimentaire naturelle des flamands roses, et ainsi impacter leur population dans les lacs africains [81]. Les études sur l'effet des bactériophages sur le microbiome des mammifères sont encore plus rares. Une étude chez des souris ayant un microbiote intestinal simplifié a révélé que certaines espèces bactériennes sont soumises à des dynamiques prédateurs-proies [82]. L’étude d’autres écosystèmes comme le microbiote intestinal humain ne sont pas aussi conclusives. Ces dynamiques pourraient avoir lieu et échapper à l’observation par les méthodes actuelles [83].
Les prophages comme « arme biologique »
Une particularité tout à fait singulière des phages tempérés est qu'ils sont capables de se maintenir dans leur hôte à l’état quiescent. Un prophage, via l’action du répresseur du cycle lytique, fournit une immunité à sa bactérie lysogène contre toute surinfection réalisée par un bactériophage similaire et cela peut affecter radicalement les interactions entre bactéries. Les bactéries lysogènes sont donc « immunisées » contre tout bactériophage du même groupe d’immunité que celui qu’elles hébergent. Ainsi, il a été proposé que des bactéries lysogènes pourraient utiliser leurs prophages comme des « armes biologiques » contre les bactéries apparentées qui utilisent la même niche écologique [84,85]. Même limitée, l’action prédatrice des bactériophages pourrait ainsi provoquer la libération partielle d’une niche écologique et permettre aux bactéries lysogènes d’augmenter en nombre. En effet, l’induction spontanée de prophages dans une très faible fraction de la population lysogénique peut déclencher une épidémie parmi les concurrents sensibles, qui deviennent alors des usines de production de bactériophages suivant le cycle lytique. Ainsi, en tuant les bactéries concurrentes, les prophages apporteraient un avantage évolutif aux génotypes bactériens « résistants » (i.e. qui portent ce prophage) et permettraient à ces bactéries lysogènes d’envahir des populations mixtes contenant des bactéries sensibles. Ce phénomène pourrait expliquer pourquoi plus de 50 % des bactéries actuellement séquencées sont lysogènes [86,87]. La généralisation de ces concepts nécessitera cependant plus d’études in vivo et dans différentes niches écologiques.
Pour un bactériophage tempéré, le choix d’entrer en lysogénie ou de réaliser un cycle lytique se fait en fonction de différents paramètres. Ainsi, le bactériophage λ privilégie le cycle lytique lors de l’infection de cellules bactériennes en croissance exponentielle, alors que la lysogénie est favorisée lorsque les bactéries infectées sont en croissance lente ou infectées par plusieurs bactériophages simultanément [88–90] . D’autres bactériophages infectant Bacillus subtilis entrent en lysogénie lorsque le ratio entre bactériophages et bactéries est fort. Cet état est décelé par un mécanisme de quorum-sensing qui détecte la concentration d’un peptide nommé «arbitrium» codé par le prophage [91]. Les facteurs influençant la balance entre les deux cycles restent à déterminer pour de nombreux modèles ainsi dans les écosystèmes naturels.
Bactériophages et brassage communautaire
Au lieu d’être bénéfique à leur hôte bactérien, les prophages peuvent lui être préjudiciables car ils sont capables d'induire la lyse de la bactérie les hébergeant. Même si l’induction spontanée d’un prophage est un événement relativement rare (fréquence de 10-4 à 10-5), un ensemble de facteurs environnementaux peut augmenter de manière importante le taux d’induction [88,92]. Cela correspond en général à des conditions délétères qui déclenchent la lysogénie permettant au prophage d’échapper à son hôte bactérien en voie de destruction. Ces conditions ne seraient pas favorables à une primo-infection lytique mais permettent une production suffisante pour infecter des hôtes naïfs. Ainsi, les prophages ont été comparés à des bombes à retardement moléculaires qui peuvent être déclenchées par des changements de salinité, des dommages à l’ADN, la température ou la présence de divers polluants dans les océans [93,94]. Dans l’intestin, plusieurs agents inducteurs peuvent potentiellement déclencher l’induction de prophages [95]. Par exemple, certains antibiotiques, tels que les quinolones ou les bêta-lactamines, déclenchent l’induction de prophages d’E. coli, de Clostridium difficile, d’Enterococcus faecalis ou encore de Staphylococcus aureus. D’autre part, le stress oxydatif, provoqué par l’inflammation est un autre inducteur des prophages dans l’intestin [96]. L’analyse de biopsies a montré que la muqueuse intestinale de patients atteints de la maladie de Crohn était 30 fois plus riche en particules pseudo-virales que celle de témoins sains [97]. L’induction des prophages dans le microbiote intestinal pourrait ainsi provoquer un «brassage de communautés» et une modification temporaire ou permanente des équilibres entre les populations bactériennes (Figure 5).
Figure 5. Le brassage communautaire lié aux souches lysogènes (source : [58]) Étape 1 : l’écosystème est à l’équilibre. Étape 2 : un stress léger survient, déclenchant l’induction des prophages des souches violettes et vertes, mais sans impact sur les souches non lysogènes. Étape 3 : les souches lysogènes ont disparu et un nouvel équilibre est atteint.
Émergence de nouvelles souches bactériennes via les prophages
La lysogénie est un facteur de transfert horizontal qui contribue considérablement à l’évolution des souches bactériennes. De nombreuses données ont montré que les prophages, de même que les prophages défectifs, affectent d’importants phénotypes bactériens (Figure 6). Les mieux caractérisés sont liés à une augmentation du pouvoir pathogène: production accrue de toxines, amélioration de la colonisation de l’hôte, augmentation de la résistance au stress oxydatif et aux sels biliaires, ou encore augmentation de la formation et la dissémination de biofilms [34,98]. Les gènes additionnels, qui ne sont pas directement impliqués dans le développement phagique, sont connus sous le nom de « morons » (ils apportent quelque chose de « plus » (more) à leur hôte). Ils comportent des signaux de transcription autonomes leur permettant d’être exprimés chez des bactéries lysogènes [99]. Fait intéressant, les études bio-informatiques suggèrent qu’il existerait de nombreux morons dans les génomes de bactériophages pour lesquels aucune fonction n’a pu être prédite. Une fois intégrés dans les génomes comme prophages, ces éléments peuvent être cooptés par l'hôte comme de nouvelles fonctions [100].
Figure 6. Les bactériophages tempérés comme vecteurs de transferts horizontaux (source : [77]). Étape 1 : deux souches de la même espèce bactérienne (verte ou bleue) cohabitent, l’une est lysogène (vert et bleu foncé), l’autre est sensible au bactériophage (vert et bleu clair, signe -). Étape 2 : les souches bactériennes sensibles sont devenues lysogènes à leur tour (vert et bleu clair, signe +), elles hébergent désormais de nouveaux gènes apportés par les bactériophages.
L’enrichissement génétique des bactéries par les phages ne se limite pas au seul génome viral : certains bactériophages peuvent parfois encapsider et transférer de larges segments d’ADN bactérien d’une souche à une autre, un processus appelé transduction généralisée. Ce processus contribue notamment à la dissémination des gènes de résistance aux antibiotiques [101]. Si le transfert de gènes de résistance aux antibiotiques reste peu documenté expérimentalement, des travaux récents ont mis en évidence que des bactéries lysogènes peuvent utiliser leurs bactériophages pour tuer des compétiteurs et, en même temps, récupérer leurs gènes de résistance aux antibiotiques [102–104]. De plus, la lyse bactérienne médiée par les bactériophages constitue également une source d’ADN libre, pouvant comporter des gènes de résistance aux antibiotiques comme c’est le cas des plasmides, pouvant être assimilée par la transformation naturelle [105].
Enfin, un dernier aspect de la contribution des bactériophages à l’émergence de souches plus virulentes est lié à leur remarquable capacité d’évolution : ils mutent et recombinent à des fréquences plus élevées que les bactéries [106]. Les bactériophages tempérés peuvent également échanger efficacement des séquences avec des éléments de bactériophages défectifs présents dans la bactérie dans laquelle ils se multiplient. De nouveaux bactériophages sont donc constamment assemblés, constitués de nouveaux assortiments de gènes. Le transfert horizontal promu par les bactériophages n’est donc pas le simple passage d’un contenu génétique donné d’un hôte à l’autre, mais il constitue un élément à part entière du processus évolutif des bactéries.
Coévolution entre phages et bactéries
Les relations prédateur-proie entre bactériophages et bactéries amènent à des dynamiques évolutives complexes. Ainsi, les bactéries ont développé plusieurs mécanismes leur permettant de résister aux infections de bactériophages. Les plus connus sont les mutations de récepteurs, les systèmes de restriction/modification qui découpent les génomes non modifiés, les systèmes d’infection abortive utilisant des mécanismes moléculaires variés , le système BREX dont la cible est le système de réplication du phage et enfin les systèmes CRISPR-Cas qui à l’aide des nucléases CAS, guidées par les ARNs détruisent les génomes phagiques entrants [107]. En réponse à ces mécanismes de défense, les bactériophages sont capables d'adopter des stratégies permettant de contourner ces résistances, e.g., en utilisant d'autres récepteurs bactériens ou des mécanismes anti-restriction ou anti-CRISPR [108–110]. Ces bactériophages augmenteront en fréquence, ce qui entraînera une chute de la population bactérienne susceptible et ainsi de suite. Ce processus co-évolutif entre hôte et parasite d’infection/résistance/contournement est nommé « course aux armements » et semble particulièrement présent dans l’interaction entre bactériophages et bactéries. Cette coévolution va générer et maintenir une grande diversité de mutations dans les populations de bactéries et de bactériophages. Cette diversité peut être représentée à l’aide de matrices de spécificité indiquant la capacité de diverses souches de bactériophages à infecter différentes souches bactériennes. La structure de ces réseaux d’interaction a des conséquences majeures sur la dynamique et le fonctionnement des écosystèmes [71].
Conclusion
L’étude des mécanismes d’infection par les bactériophages a permis la première révolution du génie génétique dans les années 70 ainsi que l’avènement de la révolution actuelle de l’édition des génomes. Ainsi, les recherches fondamentales menées sur les bactériophages en biologie moléculaire et structurale, en écologie et en biologie évolutive depuis plus de 100 ans ont conduit à des innovations majeures dans de nombreux domaines incluant les biotechnologies et la santé. Les différents aspects de la recherche actuelle sont absolument complémentaires pour comprendre le fonctionnement et l’évolution des bactériophages. Les grands défis liés à la recherche utilisant les bactériophages comme sujet d’étude concerneront bien évidemment la compréhension des mécanismes moléculaires pouvant interférer avec les approches thérapeutiques, mais également les conséquences écologiques et biogéochimiques de la prédation par les phages dans tous les écosystèmes où ils sont présents. Nul doute, que de nombreuses applications en biotechnologie et en thérapeutique continueront d’émerger à partir de ces travaux.
Remerciements
Nous remercions les membres de nos laboratoires respectifs pour leur participation aux travaux et activités du réseau Phages.fr. Nous remercions également les organismes qui financent nos activités au travers d'un Réseau Thématique Pluridisciplinaire depuis 2016: l'INSB-CNRS, l'INEE-CNRS et l'INRA. Nous tenons également à remercier chaleureusement le Dr Alain Dublanchet pour son soutien inconditionnel à nos travaux et son implication déterminante pour l’obtention du Prix de la fondation François Sommer Homme Nature 2018 attribué à notre réseau le 14 novembre 2018. Pour cette occasion, une version préliminaire de ce manuscrit a été conçue.
Note
Certains passages de cette revue sont issus du manuscrit de thèse de Nicolas Dufour http://www.theses.fr/2015USPCC259.
Références
-
Abedon ST, Thomas-Abedon C, Thomas A, Mazure H. Bacteriophage prehistory: Is or is not Hankin, 1896, a phage reference? Bacteriophage. 2011;1:174–8.
-
Hankin EH. L’action bactericide des eaux de la Jumna et du Gange sur le vibrion du cholera. Ann Inst Pasteur. 1896;10:511–23.
-
Twort FW. An investigation on the nature of ultra-microscopic viruses. The Lancet. 1915;186:1241–3.
-
d’Hérelle F. Sur un microbe invisible antagoniste des bacilles dysentériques. C R Acad Sci Paris. 1917;165:373–5.
-
d’Hérelle F. Sur le rôle du microbe filtrant bactériophage dans la dysenterie bacillaire. C R Acad Sci Paris. 1918;167:970–2.
-
d’Hérelle F. Du rôle du microbe filtrant bactériophage dans la fièvre typhoïde. C R Acad Sci Paris . 1919;168:631–4.
-
Ellis EL, Delbrück M. The growth of bacteriophage. J Gen Physiol. 1939;22:365–84.
-
Summers WC. How bacteriophage came to be used by the phage group. J Hist Biol. 1993;26:255–67.
-
Luria SE, Delbrück M. Mutations of bacteria from virus sensitivity to virus resistance. Genetics. 1943;28:491–511.
-
Hershey AD, Chase M. Independent functions of viral protein and nucleic acid in growth of bacteriophage. J Gen Physiol. 1952;36:39–56.
-
Lwoff A. Lysogeny. Bacteriol Rev. 1953;17:269–337.
-
Jacob F, Monod J. Genetic regulatory mechanisms in the synthesis of proteins. J Mol Biol. 1961;3:318–56.
-
Lederberg EM, Lederberg J. Genetic studies of lysogenicity in Escherichia coli. Genetics. 1953;38:51–64.
-
Luria SE, Anderson TF. The identification and characterization of bacteriophages with the electron microscope. Proc Natl Acad Sci U S A. 1942;28:127.
-
Sanger F, Air GM, Barrell BG, Brown NL, Coulson AR, Fiddes CA, et al. Nucleotide sequence of bacteriophage phi X174 DNA. Nature. 1977;265:687–95.
-
Fiers W, Contreras R, Duerinck F, Haegeman G, Iserentant D, Merregaert J, et al. Complete nucleotide sequence of bacteriophage MS2 RNA: primary and secondary structure of the replicase gene. Nature. 1976;260:500–7.
-
Fogg PCM, Colloms S, Rosser S, Stark M, Smith MCM. New applications for phage integrases. J Mol Biol. 2014;426:2703–16.
-
Sauer B, Whealy M, Robbins A, Enquist L. Site-specific insertion of DNA into a pseudorabies virus vector. Proc Natl Acad Sci U S A. 1987;84:9108–12.
-
Studier FW, Moffatt BA. Use of bacteriophage T7 RNA polymerase to direct selective high-level expression of cloned genes. J Mol Biol. 1986;189:113–30.
-
Walhout AJ, Temple GF, Brasch MA, Hartley JL, Lorson MA, van den Heuvel S, et al. GATEWAY recombinational cloning: application to the cloning of large numbers of open reading frames or ORFeomes. Methods Enzymol. 2000;328:575–92.
-
Eid J, Fehr A, Gray J, Luong K, Lyle J, Otto G, et al. Real-time DNA sequencing from single polymerase molecules. Science. 2009;323:133–8.
-
Doron S, Melamed S, Ofir G, Leavitt A, Lopatina A, Keren M, et al. Systematic discovery of antiphage defense systems in the microbial pangenome. Science. 2018;359.
-
Ackermann H-W. 5500 Phages examined in the electron microscope. Arch Virol. 2007;152:227–43.
-
Ackermann H-W, Kropinski AM. Curated list of prokaryote viruses with fully sequenced genomes. Res Microbiol. 2007;158:555–66.
-
Ofir G, Sorek R. Contemporary Phage Biology: From Classic Models to New Insights. Cell. 2018;172:1260–70.
-
Nash H. Site-specific recombination: integration, excision, resolution, and inversion of defined DNA segments. Escherichia coli and Salmonella Cell Mol Biol. Washington: ASM Press; 1996. p. 2363–76.
-
Gilcrease EB, Casjens SR. The genome sequence of Escherichia coli tailed phage D6 and the diversity of Enterobacteriales circular plasmid prophages. Virology. 2018;515:203–14.
-
Berngruber TW, Froissart R, Choisy M, Gandon S. Evolution of virulence in emerging epidemics. PLoS Pathog. 2013;9:e1003209.
-
Ackermann H-W, DuBow MS. Viruses of Prokaryotes: General properties of bacteriophages. CRC Press; 1987.
-
Calendar R, editor. The bacteriophages. 2nd ed. Oxford ; New York: Oxford University Press; 2006.
-
Zeng L, Golding I. Following cell-fate in E. coli after infection by phage Lambda. J Vis Exp JoVE. 2011.
-
Berngruber TW, Weissing FJ, Gandon S. Inhibition of superinfection and the evolution of viral latency. J Virol. 2010;84:10200–8.
-
Kim M-S, Bae J-W. Lysogeny is prevalent and widely distributed in the murine gut microbiota. ISME J. 2018;12:1127–41.
-
Boyd EF. Bacteriophage-encoded bacterial virulence factors and phage-pathogenicity island interactions. Adv Virus Res. 2012;82:91–118.
-
Wahl A, Battesti A, Ansaldi M. Prophages in Salmonella enterica: a driving force in reshaping the genome and physiology of their bacterial host? Mol Microbiol. 2019;111:303–16.
-
Touchon M, Moura de Sousa JA, Rocha EP. Embracing the enemy: the diversification of microbial gene repertoires by phage-mediated horizontal gene transfer. Curr Opin Microbiol. 2017;38:66–73.
-
Dowah ASA, Clokie MRJ. Review of the nature, diversity and structure of bacteriophage receptor binding proteins that target Gram-positive bacteria. Biophys Rev. 2018;
-
Bertozzi Silva J, Storms Z, Sauvageau D. Host receptors for bacteriophage adsorption. FEMS Microbiol Lett. 2016;363.
-
Pires DP, Oliveira H, Melo LDR, Sillankorva S, Azeredo J. Bacteriophage-encoded depolymerases: their diversity and biotechnological applications. Appl Microbiol Biotechnol. 2016;100:2141–51.
-
Hayes S, Vincentelli R, Mahony J, Nauta A, Ramond L, Lugli GA, et al. Functional carbohydrate binding modules identified in evolved dits from siphophages infecting various Gram-positive bacteria. Mol Microbiol. 2018;110:777–95.
-
Georgopoulos C. Toothpicks, serendipity and the emergence of the Escherichia coli DnaK (Hsp70) and GroEL (Hsp60) chaperone machines. Genetics. 2006;174:1699–707.
-
Aksyuk AA, Rossmann MG. Bacteriophage assembly. Viruses. 2011;3:172–203.
-
Dubochet J. Cryo-EM–the first thirty years. J Microsc. 2012;245:221–4.
-
Lepault J, Dubochet J, Baschong W, Kellenberger E. Organization of double-stranded DNA in bacteriophages: a study by cryo-electron microscopy of vitrified samples. EMBO J. 1987;6:1507–12.
-
Oliveira L, Tavares P, Alonso JC. Headful DNA packaging: bacteriophage SPP1 as a model system. Virus Res. 2013;173:247–59.
-
Ponchon L, Mangenot S, Boulanger P, Letellier L. Encapsidation and transfer of phage DNA into host cells: from in vivo to single particles studies. Biochim Biophys Acta. 2005;1724:255–61.
-
Preux O, Durand D, Huet A, Conway JF, Bertin A, Boulogne C, et al. A two-state cooperative expansion converts the procapsid shell of bacteriophage T5 into a highly stable capsid isomorphous to the final virion head. J Mol Biol. 2013;425:1999–2014.
-
Tavares P, Zinn-Justin S, Orlova EV. Genome gating in tailed bacteriophage capsids. Adv Exp Med Biol. 2012;726:585–600.
-
Vernhes E, Renouard M, Gilquin B, Cuniasse P, Durand D, England P, et al. High affinity anchoring of the decoration protein pb10 onto the bacteriophage T5 capsid. Sci Rep. 2017;7:41662.
-
Bauer DW, Evilevitch A. Influence of Internal DNA Pressure on Stability and Infectivity of Phage λ. J Mol Biol. 2015;427:3189–200.
-
Taylor NMI, Prokhorov NS, Guerrero-Ferreira RC, Shneider MM, Browning C, Goldie KN, et al. Structure of the T4 baseplate and its function in triggering sheath contraction. Nature. 2016;533:346–52.
-
Arnaud C-A, Effantin G, Vivès C, Engilberge S, Bacia M, Boulanger P, et al. Bacteriophage T5 tail tube structure suggests a trigger mechanism for Siphoviridae DNA ejection. Nat Commun. 2017;8:1953.
-
Legrand P, Collins B, Blangy S, Murphy J, Spinelli S, Gutierrez C, et al. The Atomic Structure of the Phage Tuc2009 Baseplate Tripod Suggests that Host Recognition Involves Two Different Carbohydrate Binding Modules. mBio. 2016;7:e01781-01715.
-
Yap ML, Rossmann MG. Structure and function of bacteriophage T4. Future Microbiol. 2014;9:1319–27.
-
Wikoff WR, Liljas L, Duda RL, Tsuruta H, Hendrix RW, Johnson JE. Topologically linked protein rings in the bacteriophage HK97 capsid. Science. 2000;289:2129–33.
-
Hu B, Margolin W, Molineux IJ, Liu J. The bacteriophage t7 virion undergoes extensive structural remodeling during infection. Science. 2013;339:576–9.
-
Hu B, Margolin W, Molineux IJ, Liu J. Structural remodeling of bacteriophage T4 and host membranes during infection initiation. Proc Natl Acad Sci U S A. 2015;112:4919-4928.
-
McNulty R, Cardone G, Gilcrease EB, Baker TS, Casjens SR, Johnson JE. Cryo-EM Elucidation of the Structure of Bacteriophage P22 Virions after Genome Release. Biophys J. 2018;114:1295–301.
-
Wang C, Tu J, Liu J, Molineux IJ. Structural dynamics of bacteriophage P22 infection initiation revealed by cryo-electron tomography. Nat Microbiol. 2019;4:1049.
-
Smith GP. Filamentous fusion phage: novel expression vectors that display cloned antigens on the virion surface. Science. 1985;228:1315–7.
-
Peralta B, Gil-Carton D, Castaño-Díez D, Bertin A, Boulogne C, Oksanen HM, et al. Mechanism of membranous tunnelling nanotube formation in viral genome delivery. PLoS Biol. 2013;11:e1001667.
-
Cvirkaite-Krupovic V, Krupovic M, Daugelavicius R, Bamford DH. Calcium ion-dependent entry of the membrane-containing bacteriophage PM2 into its Pseudoalteromonas host. Virology. 2010;405:120–8.
-
Sun L, Young LN, Zhang X, Boudko SP, Fokine A, Zbornik E, et al. Icosahedral bacteriophage ΦX174 forms a tail for DNA transport during infection. Nature. 2014;505:432–5.
-
Vinga I, São-José C, Tavares P, Santos MA. Bacteriophage entry in the host cell. Mod Bacteriophage Biol Biotechnol. Ed. G. Wegrzyn. Research Signpost; 2006. p. 163–203.
-
Cvirkaite-Krupovic V, Poranen MM, Bamford DH. Phospholipids act as secondary receptor during the entry of the enveloped, double-stranded RNA bacteriophage phi6. J Gen Virol. 2010;91:2116–20.
-
Samire P, Serrano B, Duche D, Lemarie E, Lloubes R, Houot L. Decoupling filamentous phage uptake and energy of the TolQRA motor in Escherichia coli. J Bacteriol. 2019;
-
Dominguez-Medina S, Fostner S, Defoort M, Sansa M, Stark A-K, Halim MA, et al. Neutral mass spectrometry of virus capsids above 100 megadaltons with nanomechanical resonators. Science. 2018;362:918–22.
-
Suttle CA. Marine viruses–major players in the global ecosystem. Nat Rev Microbiol. 2007;5:801–12.
-
Shkoporov AN, Hill C. Bacteriophages of the Human Gut: The “Known Unknown” of the Microbiome. Cell Host Microbe. 2019;25:195–209.
-
LePage DP, Metcalf JA, Bordenstein SR, On J, Perlmutter JI, Shropshire JD, et al. Prophage WO genes recapitulate and enhance Wolbachia-induced cytoplasmic incompatibility. Nature. 2017;543:243–7.
-
Koskella B. New approaches to characterizing bacteria-phage interactions in microbial communities and microbiomes. Environ Microbiol Rep. 2019;11:15–6.
-
Koskella B, Taylor TB. Multifaceted Impacts of Bacteriophages in the Plant Microbiome. Annu Rev Phytopathol. 2018;56:361–80.
-
Edlin G, Lin L, Bitner R. Reproductive fitness of P1, P2, and Mu lysogens of Escherichia coli. J Virol. 1977;21:560–4.
-
Wang X, Kim Y, Ma Q, Hong SH, Pokusaeva K, Sturino JM, et al. Cryptic prophages help bacteria cope with adverse environments. Nat Commun. 2010;1:147.
-
Weinbauer MG, Rassoulzadegan F. Are viruses driving microbial diversification and diversity? Environ Microbiol. 2004;6:1–11.
-
Díaz-Muñoz SL, Koskella B. Bacteria-phage interactions in natural environments. Adv Appl Microbiol. 2014;89:135–83.
-
De Paepe M, Leclerc M, Tinsley CR, Petit M-A. Bacteriophages: an underestimated role in human and animal health? Front Cell Infect Microbiol. 2014;4:39.
-
Winter C, Bouvier T, Weinbauer MG, Thingstad TF. Trade-offs between competition and defense specialists among unicellular planktonic organisms: the “killing the winner” hypothesis revisited. Microbiol Mol Biol Rev MMBR. 2010;74:42–57.
-
Faruque SM, Naser IB, Islam MJ, Faruque ASG, Ghosh AN, Nair GB, et al. Seasonal epidemics of cholera inversely correlate with the prevalence of environmental cholera phages. Proc Natl Acad Sci U S A. 2005;102:1702–7.
-
Marston MF, Sallee JL. Genetic diversity and temporal variation in the cyanophage community infecting marine Synechococcus species in Rhode Island’s coastal waters. Appl Environ Microbiol. 2003;69:4639–47.
-
Peduzzi P, Gruber M, Gruber M, Schagerl M. The virus’s tooth: cyanophages affect an African flamingo population in a bottom-up cascade. ISME J. 2014;8:1346–51.
-
Reyes A, Wu M, McNulty NP, Rohwer FL, Gordon JI. Gnotobiotic mouse model of phage-bacterial host dynamics in the human gut. Proc Natl Acad Sci U S A. 2013;110:20236–41.
-
De Sordi L, Lourenço M, Debarbieux L. The Battle Within: Interactions of Bacteriophages and Bacteria in the Gastrointestinal Tract. Cell Host Microbe. 2019;25:210–8.
-
Bossi L, Fuentes JA, Mora G, Figueroa-Bossi N. Prophage Contribution to Bacterial Population Dynamics. J Bacteriol. 2003;185:6467–71.
-
Brown SP, Le Chat L, De Paepe M, Taddei F. Ecology of microbial invasions: amplification allows virus carriers to invade more rapidly when rare. Curr Biol CB. 2006;16:2048–52.
-
Roux S, Hallam SJ, Woyke T, Sullivan MB. Viral dark matter and virus-host interactions resolved from publicly available microbial genomes. eLife. 2015;4:e08490.
-
Touchon M, Bernheim A, Rocha EP. Genetic and life-history traits associated with the distribution of prophages in bacteria. ISME J. 2016;10:2744–54.
-
Gandon S. Why Be Temperate: Lessons from Bacteriophage λ. Trends Microbiol. 2016;24:356–65.
-
Kourilsky P, Knapp A. Lysogenization by bacteriophage lambda. III. Multiplicity dependent phenomena occuring upon infection by lambda. Biochimie. 1974;56:1517–23.
-
Zeng L, Skinner SO, Zong C, Sippy J, Feiss M, Golding I. Decision making at a subcellular level determines the outcome of bacteriophage infection. Cell. 2010;141:682–91.
-
Erez Z, Steinberger-Levy I, Shamir M, Doron S, Stokar-Avihail A, Peleg Y, et al. Communication between viruses guides lysis-lysogeny decisions. Nature. 2017;541:488–93.
-
Kim S, Ryu K, Biswas D, Ahn J. Survival, prophage induction, and invasive properties of lysogenic Salmonella Typhimurium exposed to simulated gastrointestinal conditions. Arch Microbiol. 2014;196:655–9.
-
Paul JH. Prophages in marine bacteria: dangerous molecular time bombs or the key to survival in the seas? ISME J. 2008;2:579–89.
-
Shan J, Korbsrisate S, Withatanung P, Adler NL, Clokie MRJ, Galyov EE. Temperature dependent bacteriophages of a tropical bacterial pathogen. Evol Genomic Microbiol. 2014;5:599.
-
De Paepe M, Tournier L, Moncaut E, Son O, Langella P, Petit M-A. Carriage of λ latent virus is costly for its bacterial host due to frequent reactivation in monoxenic mouse intestine. PLoS Genet. 2016;12:e1005861.
-
Diard M, Bakkeren E, Cornuault JK, Moor K, Hausmann A, Sellin ME, et al. Inflammation boosts bacteriophage transfer between Salmonella spp. Science. 2017;355:1211–5.
-
Lepage P, Leclerc MC, Joossens M, Mondot S, Blottière HM, Raes J, et al. A metagenomic insight into our gut’s microbiome. Gut. 2013;62:146–58.
-
Brüssow H, Canchaya C, Hardt W-D. Phages and the evolution of bacterial pathogens: from genomic rearrangements to lysogenic conversion. Microbiol Mol Biol Rev MMBR. 2004;68:560–602.
-
Cumby N, Davidson AR, Maxwell KL. The moron comes of age. Bacteriophage. 2012;2:225–8.
-
Bobay L-M, Touchon M, Rocha EPC. Pervasive domestication of defective prophages by bacteria. Proc Natl Acad Sci U S A. 2014;111:12127–32.
-
Salmond GPC, Fineran PC. A century of the phage: past, present and future. Nat Rev Microbiol. 2015;13:777–86.
-
Enault F, Briet A, Bouteille L, Roux S, Sullivan MB, Petit M-A. Phages rarely encode antibiotic resistance genes: a cautionary tale for virome analyses. ISME J. 2017;11:237–47.
-
Haaber J, Leisner JJ, Cohn MT, Catalan-Moreno A, Nielsen JB, Westh H, et al. Bacterial viruses enable their host to acquire antibiotic resistance genes from neighbouring cells. Nat Commun 2016;7.
-
Modi SR, Lee HH, Spina CS, Collins JJ. Antibiotic treatment expands the resistance reservoir and ecological network of the phage metagenome. Nature. 2013;499:219–22.
-
Keen EC, Bliskovsky VV, Malagon F, Baker JD, Prince JS, Klaus JS, et al. Novel “Superspreader” Bacteriophages Promote Horizontal Gene Transfer by Transformation. mBio. 2017;8:e02115-16.
-
De Paepe M, Hutinet G, Son O, Amarir-Bouhram J, Schbath S, Petit M-A. Temperate phages acquire DNA from defective prophages by relaxed homologous recombination: the role of Rad52-like recombinases. PLoS Genet. 2014;10:e1004181.
-
Labrie SJ, Samson JE, Moineau S. Bacteriophage resistance mechanisms. Nat Rev Microbiol. 2010;8:317–27.
-
Meyer JR, Dobias DT, Medina SJ, Servilio L, Gupta A, Lenski RE. Ecological speciation of bacteriophage lambda in allopatry and sympatry. Science. 2016;354:1301–4.
-
Samson JE, Magadán AH, Sabri M, Moineau S. Revenge of the phages: defeating bacterial defences. Nat Rev Microbiol. 2013;11:675–87.
-
Pawluk A, Davidson AR, Maxwell KL. Anti-CRISPR: discovery, mechanism and function. Nat Rev Microbiol. 2018;16:12–7.